SAPROBITY IDENTIFICATION OF HYDROBIONT SPECIES OF VERHNIY KABAN LAKE OF KAZAN BY 18S rRNA MARKER GENE
JOURNAL: «SCIENTIFIC NOTES OF V.I. VERNADSKY CRIMEAN FEDERAL UNIVERSITY. Biology. Chemistry» Volume 6 (72), №4, 2020
Publication text (PDF):Download
UDK: 577;574
AUTHOR AND PUBLICATION INFORMATION
AUTHORS:
Sverdrup A. E., Kazan Federal University, Kazan, Russia
Frolova L. L., Kazan Federal University, Kazan, Russia
TYPE:Article
DOI:https://doi.org/10.37279/2413-1725-2020-6-4-127-142
PAGES:from 127 to 142
STATUS:Published
LANGUAGE:Russian
KEYWORDS: molecular phylogeny, 18S rRNA marker gene, hydrobionts, freshwater reservoirs, saprobity, Lake Verhniy Kaban.
ABSTRACT (ENGLISH):
The paper presents the results of a study of the hypothetical evolution of unicellular hydrobionts from Verhniy Kaban Lake of Kazan by 18S rRNA marker gene with aim of identifying species saprobity based on molecular phylogenetic analysis.
Verhniy Kaban Lake is an isolated freshwater reservoir. Its coastline is relatively smooth except for the northern end, which forms swampy and overgrown bay. To the West of lake an urban settlement is situated and to the East are highway and garden plots. Lake length is about 1 km, average and maximum widths are 245 m and 370 m respectively. Deepest point is 15 m; average depth is 8 m; lake area – 25 hectares.
Quality of lake water is classified as polluted. One of methods used for ecological assessment of water reservoirs is the bioindication method based on ability of indicator species to survive in water of certain pollution degree. At present, freshwater hydrobiont species from V.Sladechek’s list (1973) are used as indicators. Indicator species lists expand very slowly due to the long process of experimental observations. At the same time, price drop of the next-generation sequencing technologies allows to identify more organisms and run molecular phylogenetic analysis with aim of identifying species saprobity based on study of hypothetic evolution of hydrobionts by marker genes including 18S rRNA. The advantage of using 18S rRNA/16S rRNA ribosomal genes is that they are present in all organisms. Ribosomal genes are ones of the most conservative genes. Therefore, the systematic position of the organism and the time of divergence with closely related species can be determined via analysis of similarities and differences in rRNA sequences.
Molecular phylogenetic trees were constructed by the neighbour joining (NJ) and maximal parsimony (MP) methods for Verhniy Kaban Lake hydrobionts identified with next-generation sequencing method (Illumina) by 18S rRNA marker gene (SRR7510986, SRR7465374, SRR7516513). Phylogenetic analysis of hydrobionts of Verhniy Kaban Lake by 18S rRNA marker gene revealed 12 clusters with a high bootstrap value
(68–100 %), containing 12 existing indicator species of hydrobionts and 13 unicellular algae and ciliates – indicator species of different saprobity that did not previously have the status of bioindicators – algae: Diplosphaera mucosa (o-b-saprobity), Mucidosphaerium pulchellum (p-a-saprobity), Dinobryon pediforme (b-mesosaprobity), Gomphoneis minuta
(b-mesosaprobity), Nitzschia amphibia (o-b-saprobity), Rhabdonema adriaticum
(o-b-saprobity), Synura mammillosa (b-o-saprobity), Cryptomonas ozolini
(b-mesosaprobity); ciliates: Astylozoon enriquesi (b-mesosaprobity), Blepharocorys curvigula (p-saprobity), Enchelys gasterosteus (p-saprobity), Monocoronella carnea
(a-b-mesosaprobity), Plagiopogon loricatus (b-a-mesosaprobity).
Most identified indicator species group near b-mesosaprobity. This indicates ecological state of Verhniy Kaban Lake as polluted by ecological assessments.
ВВЕДЕНИЕ
Озера Кабан расположены в черте крупного промышленного города Казани и постоянно испытывают антропогенную нагрузку. Они представляют собой водную систему, состоящую из трёх крупных городских водоёмов – Верхний Кабан, Средний Кабан и Нижний Кабан. Озеро Верхний Кабан – обособленный водоём, который расположен в 1,3 км от озера Средний Кабан, не соединяется с остальными водоёмами и обладает наименьшими размерами. Береговая линия сравнительно ровная, за исключением северного конца, образующего узкий залив, частично заболоченный и зарастающий. На западном берегу озера располагается городской посёлок, на восточном берегу – автодорога и далее, на склоне, садовые участки. Длина озера составляет около 1 км, средняя ширина 245 м, максимальная ширина – 370 м. Наибольшая глубина озера – 15 м; средняя глубина – 8 м; площадь озера – 25 га [1].
По санитарно-экологическим показателям озера Кабан относятся к загрязнённым. Одним из методов, используемых для оценки экологического состояния водоёмов, является метод биоиндикации, основанный на способности индикаторных видов организмов выживать в воде с той или иной определённой степенью загрязнения. В настоящее время в качестве индикаторов используются виды пресноводных гидробионтов из списка В. Сладечека (1973) [2]. Списки видов-индикаторов пополняются крайне медленно из-за длительности процесса проведения экспериментальных наблюдений. В то же время снижение стоимости технологий секвенирования нового поколения с высокой пропускной способностью позволяет идентифицировать большое количество организмов [3–5] и проводить молекулярный филогенетический анализ с целью выявления принадлежности видов к зонам сапробности на основе исследования гипотетического хода эволюции видов гидробионтов по маркерным генам [6], в том числе по гену 18S рРНК. Преимуществом исследования по рибосомным генам 18S рРНК/16S рРНК является их присутствие во всех организмах. Гены рРНК – одни из наиболее консервативных генов. Поэтому систематическое положение организма и время расхождения с близкими видами могут быть определены на основании анализа сходств и различий в последовательностях рРНК [7].
Выявление бóльшего количества видов-индикаторов значительно повысит достоверность оценки качества воды водоёмов.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использованы современные методы молекулярной биологии, биоинформатики и молекулярной филогении.
Поиск и множественное выравнивание нуклеотидных последовательностей маркерного гена 18S рРНК гидробионтов проведены с использованием онлайн-программ на сайте NCBI (https://www.ncbi.nlm.nih.gov/), реконструкция и визуализация молекулярных филогенетических деревьев проведены с помощью программы MEGA-X (Molecular Evolutionary Genetics Analysis, http://www.megasoftware.net/).
Молекулярные филогенетические деревья построены методами присоединения соседей (NJ) [8, 9] и максимальной экономии (MP) [10, 11] для гидробионтов озера Верхний Кабан, идентификация которых была проведена по методу секвенирования нового поколения (Illumina) с использованием маркерного гена 18S рРНК (SRR7510986, SRR7465374, SRR7516513). Бутстреп-значения приведены рядом с узлами обобщённого дерева [12]. В таблице 1 рядом с названием вида указана его сапробность из списка В. Сладечека (1973), приведён идентификационный номер его нуклеотидной последовательности 18S рРНК в международной базе данных GenBank.
Таблица 1
Идентификационные номера нуклеотидных последовательностей 18S рРНК видов гидробионтов в базе данных GenBank
| S* | Вид | NCBI ID | S | Вид | NCBI ID |
| Achlya bisexualis | 166300 | Lagerheimia longiseta | 442571948 | ||
| Achnanthidium minutissimum | 789408363 | Lagynion scherffelii | 5257240 | ||
| p | Acineria incurvata | 645759758 | Licmophora abbreviata | 924436425 | |
| Actinotaenium phymatosporum | 23304137 | Licmophora grandis | 126116676 | ||
| Amphora laevissima | 612408392 | Lindernia crustacea | 284975304 | ||
| o-b | Amphora ovalis | 612408395 | Makinoella tosaensis | 824632969 | |
| Apokeronopsis crassa | 404425838 | Meyerella planktonica | 28883446 | ||
| o-b | Asterionella formosa | 370991436 | Monocoronella carnea | 224579099 | |
| Astragalus variabilis | 751813766 | Mucidosphaerium pulchellum | 37362085 | ||
| Astylozoon enriquesi | 17223768 | Muriella terrestris | 3721581 | ||
| Bacillaria paxillifer | 959096469 | Myoschiston cf.duplicatum | 381149199 | ||
| Bicosoeca kenaiensis | 749396754 | Nassula labiata | 558697254 | ||
| Blepharocorys curvigula | 269246308 | Navicula cf.normaloides | 1005406241 | ||
| o-b | Botryococcus braunii | 225903793 | Navicula cryptotenelloides | 952988073 | |
| Bryometopus triquetrus | 675304917 | Neobodo borokensis | 930722314 | ||
| Cassytha filiformis | 523916672 | Nitzschia amphibia | 56398975 | ||
| Chamabainia cuspidata | 557162315 | o-b | Nitzschia fonticola | 58416170 | |
| Chilodonella acuta | 588494198 | Paranophrys magna | 430737142 | ||
| Chilodonella parauncinata | 693735214 | Pavlova granifera | 330423569 | ||
| p-a | Chlorella vulgaris | 1237896213 | Physaria ludoviciana | 984915404 | |
| Chromophyton vischeri | 281371119 | Phytophthora porri | 403181314 | ||
| Coccolithus braarudii | 32879327 | Phytophthora sojae | 53693098 | ||
| b | Cocconeis placentula | 329343421 | Placus salinus | 558697259 | |
| Coleochaete scutata | 18289 | Plagiopogon loricatus | 514076798 | ||
| b-a | Coleps hirtus | 4090657 | Plagioselmis nannoplanctica | 215262414 | |
| Croton texensis | 984915385 | Porpostoma notata | 306032968 | ||
| b | Cryptomonas curvata | 1003585960 | Protocyclidium sinica | 514076830 | |
| Cryptomonas ozolini | 529274331 | Rhabdonema adriaticum | 555823261 | ||
| Cucumis melo | 13876599 | Sartidia perrieri | 1001184035 | ||
| Cyathea poeppigii | 109138938 | Siluania monomastiga | 3335210 | ||
| Cyclonexis annularis | 5257246 | Sorodiplophrys stercorea | 1000926255 | ||
| Cyclotella choctawhatcheeana | 168145946 | Storeatula major | 1777916 | ||
| b-a | Cymatopleura solea | 906411159 | Synura mammillosa | 2058487 | |
| b | Cymbella cistula | 725655009 | b | Synura petersenii | 2058488 |
| Dexitrichides pangi | 28974302 | b-o | Synura spinosa | 859809142 | |
| b | Dinobryon divergens | 1210424080 | Telotrochidium matiense | 157367079 | |
| Dinobryon pediforme | 728802316 | Tessellaria volvocina | 2058491 | ||
| Diplosphaera mucosa | 664686831 | o-b | Tintinnidium fluviatile | 388540836 | |
| Emiliania huxleyi | 514253898 | Trithigmostoma cucullulus | 290581700 | ||
| Enchelys gasterosteus | 324499387 | Uroglenopsis americana | 5257242 | ||
| Epistylis chlorelligerum | 699360684 | Urosolenia eriensis | 329343406 | ||
| Eunotia minor | 789408372 | Urosomoida agilis | 674781142 | ||
| Exanthemachrysis gayraliae | 99907146 | a-b | Urostyla grandis | 822576503 | |
| Glaucocystis nostochinearum | 683567 | Vampyrella lateritia | 378734995 | ||
| Gomphoneis minuta | 725655038 | p | Vorticella aequilata | 363412175 | |
| Goniomonas avonlea | 411170591 | b | Vorticella mayeri | 1002824137 | |
| Hibberdia magna | 174830 | Zoothamnium pluma | 110294735 | ||
| Humidophila australis | 694880111 | Zygnema circumcarinatum | 669688778 | ||
| Hyalosynedra cf.laevigata | 126116673 | Lewinskya sordidum | 109138912 | ||
| Kleinstyla dorsicirrata | 471272487 | *S – сапробность по В.Сладечеку: о-олиго-, b-бетамезо-, a-альфамезо-, р-поли-сапробы | |||
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Молекулярные филогенетические деревья построены двумя методами – присоединения соседей (NJ) и максимальной экономии (MP) – для гидробионтов озера Верхний Кабан, идентификация которых была проведена по методу секвенирования нового поколения (Illumina) с использованием маркерного гена
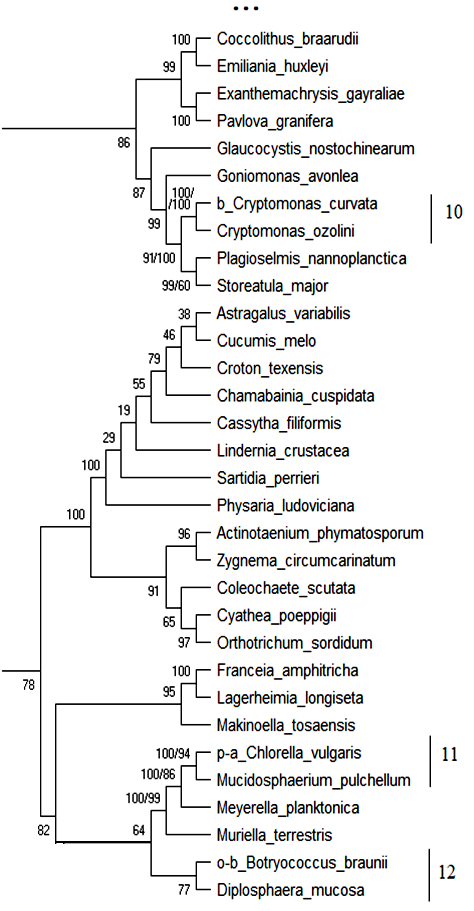
18S рРНК. Обобщённое филогенетическое дерево по маркерному гену 18S рРНК гидробионтов озера Верхний Кабан приведено на рис. 1. Бутстреп-значения приведены рядом с узлами дерева.
Как видно из рис. 1, на обобщённом молекулярном филогенетическом дереве выявлено 12 кластеров с высоким бутстреп-значением, содержащих индикаторные виды гидробионтов и виды-индикаторы разных зон сапробности, не имевших ранее статус индикаторных видов:
Кластер 1: Вид Vorticella mayeri – b-индикатор сгруппирован с неиндикаторным видом Astylozoon enriquesi с высоким бутстреп-значением 100 %. В табл. 2 приведено таксономическое положение сгруппированных организмов.
Таблица 2
Таксономическое положение сгруппированных гидробионтов
Vorticella mayeri и Astylozoon enriquesi
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Ciliophora | Ciliophora |
| Class | Oligohymenophorea | Oligohymenophorea |
| Order | Sessilida | Sessilida |
| Family | Vorticellidae | Astylozoidae |
| Genus, Species | Vorticella mayeri | Astylozoon enriquesi |
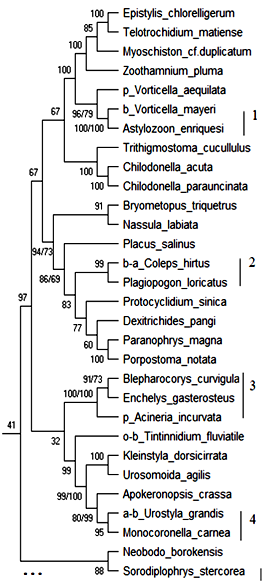
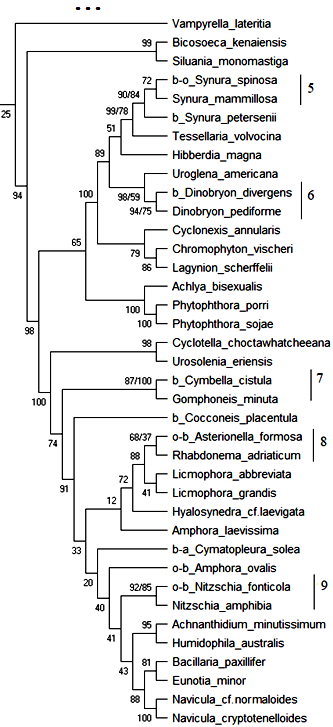
Рис. 1. Обобщённое филогенетическое дерево по маркерному гену 18S рРНК гидробионтов озера Верхний Кабан (NJ/MP).
Продолжение рис. 1.
Как видно из табл. 2 кластер 1 содержит организмы, близкие по таксономическому положению, и вид Astylozoon enriquesi может рассматриваться как индикатор b–мезосапробности с вероятностью 100 %.
Кластер 2: Вид Coleps hirtus – b–a–индикатор сгруппирован с неиндикаторным видом Plagiopogon loricatus с высоким бутстреп-значением 99 %. В табл. 3 приведено таксономическое положение сгруппированных организмов.
Таблица 3
Таксономическое положение сгруппированных гидробионтов
Coleps hirtus и Plagiopogon loricatus
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Ciliophora | Ciliophora |
| Class | Prostomatea | Prostomatea |
| Order | Prorodontida | Prorodontida |
| Family | Colepidae | Colepidae |
| Genus, Species | Coleps hirtus | Plagiopogon loricatus |
Как видно из табл. 3 кластер 2 содержит организмы, близкие по таксономическому положению, и вид Plagiopogon loricatus может рассматриваться как индикатор b–a–мезосапробности с вероятностью 99 %.
Кластер 3: Вид Acineria incurvata – p–индикатор сгруппирован с неиндикаторными видами Blepharocorys curvigula и Enchelys gasterosteus с высоким бутстреп-знчением 100 %. В табл. 4 приведено таксономическое положение сгруппированных организмов.
Таблица 4
Таксономическое положение сгруппированных гидробионтов
Acineria incurvata, Blepharocorys curvigula и Enchelys gasterosteus
| Domain | Eukaryota | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista | Chromista |
| Phylum | Ciliophora | Ciliophora | Ciliophora |
| Class | Litostomatea | Litostomatea | Litostomatea |
| Order | Pleurostomatida | Entodiniomorphida | Haptorida |
| Family | Litonotidae | Blepharocorythidae | Enchelyidae |
| Genus, Species | Acineria incurvata | Blepharocorys curvigula | Enchelys gasterosteus |
Как видно из табл. 4 кластер 3 содержит организмы близкие по таксономическому положению до уровня Class, и виды Blepharocorys curvigula и Enchelys gasterosteus могут рассматриваться как индикаторы p–сапробности с вероятностью 100 %.
Кластер 4: Вид Urostyla grandis – a–b-индикатор сгруппирован с неиндикаторным видом Monocoronella carnea с высоким бутстреп-значением 95 %. В табл. 5 приведено таксономическое положение сгруппированных организмов.
Таблица 5
Таксономическое положение сгруппированных гидробионтов
Urostyla grandis и Monocoronella carnea
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Ciliophora | Ciliophora |
| Class | Spirotrichea | Spirotrichea |
| Order | Urostylida | Urostylida |
| Family | Urostylidae | Urostylida incertae sedis |
| Genus, Species | Urostyla grandis | Monocoronella carnea |
Как видно из табл. 5 кластер 4 содержит организмы близкие по таксономическому положению, и вид Monocoronella carnea может рассматриваться как индикатор a–b–мезосапробности с вероятностью 95 %.
Кластер 5: Вид Synura spinosa – b–o–индикатор сгруппирован с неиндикаторным видом Synura mammillosa с высоким бутстреп-значением 72 %. В табл. 6 приведено таксономическое положение сгруппированных организмов.
Таблица 6
Таксономическое положение сгруппированных гидробионтов
Synura spinosa и Synura mammillosa
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Heterokonta | Heterokonta |
| Class | Synurophyceae | Synurophyceae |
| Order | Synurales | Synurales |
| Family | Mallomonadaceae | Mallomonadaceae |
| Genus, Species | Synura spinosa | Synura mammillosa |
Как видно из табл. 6 кластер 5 содержит организмы, близкие по таксономическому положению, и вид Synura mammillosa может рассматриваться как индикатор b–o–сапробности с вероятностью 72 %.
Кластер 6: Вид Dinobryon divergens – b–индикатор сгруппирован с неиндикаторным видом Dinobryon pediforme с высоким бутстреп-значением 94 %. В табл. 7 приведено таксономическое положение сгруппированных организмов.
Таблица 7
Таксономическое положение сгруппированных гидробионтов
Dinobryon divergens и Dinobryon pediforme
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Ochrophyta | Ochrophyta |
| Class | Chrysophyceae | Chrysophyceae |
| Order | Chromulinales | Chromulinales |
| Family | Dinobryaceae | Dinobryaceae |
| Genus, Species | Dinobryon divergens | Dinobryon pediforme |
Как видно из табл. 7 кластер 6 содержит организмы, близкие по таксономическому положению, и вид Dinobryon pediforme может рассматриваться как индикатор b–мезосапробности с вероятностью 94 %.
Кластер 7: Вид Cymbella cistulla – b-индикатор сгруппирован с неиндикаторным видом Gomphoneis minuta с высоким бутстреп-значением 100 %. В табл. 8 приведено таксономическое положение сгруппированных организмов.
Таблица 8
Таксономическое положение сгруппированных гидробионтов
Cymbella cistulla и Gomphoneis minuta
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Ochrophyta | Ochrophyta |
| Class | Bacillariophyceae | Bacillariophyceae |
| Order | Cymbellales | Cymbellales |
| Family | Cymbellaceae | Gomphonemataceae |
| Genus, Species | Cymbella cistula | Gomphoneis minuta |
Как видно из табл. 8 кластер 7 содержит организмы, близкие по таксономическому положению, и вид Gomphoneis minuta может рассматриваться как индикатор b–мезосапробности с вероятностью 100 %.
Кластер 8: Вид Asterionella formosa – o–b-индикатор сгруппирован с неиндикаторным видом Rhabdonema adriaticum с бутстреп-значением 68 %. В табл. 9 приведено таксономическое положение сгруппированных организмов.
Таблица 9
Таксономическое положение сгруппированных гидробионтов
Asterionella formosa и Rhabdonema adriaticum
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Ochrophyta | Ochrophyta |
| Class | Fragilariophyceae | Bacillariophyceae |
| Order | Fragilariales | Rhabdonematales |
| Family | Fragilariaceae | Rhabdonemataceae |
| Genus, Species | Asterionella formosa | Rhabdonema adriaticum |
Как видно из табл. 9 кластер 8 содержит организмы, близкие по таксономическому положению до уровня Phylum, и вид Rhabdonema adriaticum может рассматриваться как индикатор o–b-сапробности с вероятностью 68 %.
Кластер 9: Вид Nitzschia fonticola – o–b-индикатор сгруппирован с неиндикаторным видом Nitzschia amphibia с высоким бутстреп-значением 92 %. В табл. 10 приведено таксономическое положение сгруппированных организмов.
Таблица 10
Таксономическое положение сгруппированных гидробионтов
Nitzschia fonticola и Nitzschia amphibia
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Ochrophyta | Ochrophyta |
| Class | Bacillariophyceae | Bacillariophyceae |
| Order | Bacillariales | Bacillariales |
| Family | Bacillariaceae | Bacillariaceae |
| Genus, Species | Nitzschia fonticola | Nitzschia amphibia |
Как видно из табл. 10 кластер 9 содержит организмы, близкие по таксономическому положению, и вид Nitzschia amphibia может рассматриваться как индикатор o–b–сапробности с вероятностью 92 %.
Кластер 10: Вид Cryptomonas curvata – b–индикатор сгруппирован с неиндикаторным видом Cryptomonas ozolini с высоким бутстреп-значением 100 %. В табл. 11 приведено таксономическое положение сгруппированных организмов.
Таблица 11
Таксономическое положение сгруппированных гидробионтов
Cryptomonas curvata и Cryptomonas ozolini
| Domain | Eukaryota | Eukaryota |
| Kingdom | Chromista | Chromista |
| Phylum | Cryptophyta | Cryptophyta |
| Class | Cryptophyceae | Cryptophyceae |
| Order | Cryptomonadales | Cryptomonadales |
| Family | Cryptomonadaceae | Cryptomonadaceae |
| Genus, Species | Cryptomonas curvata | Cryptomonas ozolini |
Как видно из табл. 11 кластер 10 содержит организмы, близкие по таксономическому положению, и вид Cryptomonas ozolini может рассматриваться как индикатор b–мезосапробности с вероятностью 100 %.
Кластер 11: Вид Chlorella vulgaris – p–a–индикатор сгруппирован с неиндикаторным видом Mucidosphaerium pulchellum с высоким бутстреп-значением 100 %. В табл. 12 приведено таксономическое положение сгруппированных организмов.
Таблица 12
Таксономическое положение сгруппированных гидробионтов
Chlorella vulgaris и Mucidosphaerium pulchellum
| Domain | Eukaryota | Eukaryota |
| Kingdom | Plantae | Plantae |
| Phylum | Chlorophyta | Chlorophyta |
| Class | Trebouxiophyceae | Trebouxiophyceae |
| Order | Chlorellales | Chlorellales |
| Family | Chlorellaceae | Chlorellaceae |
| Genus, Species | Chlorella vulgaris | Mucidosphaerium pulchellum |
Как видно из табл. 12 кластер 11 содержит организмы близкие по таксономическому положению, и вид Mucidosphaerium pulchellum может рассматриваться как p–a–индикатором с вероятностью 100 %.
Кластер 12: Вид Botryococcus brauni – o–b–индикатор сгруппирован с неиндикаторным видом Diplosphaera mucosa с высоким бутстреп-значением 77 %. В табл. 13 приведено таксономическое положение сгруппированных организмов.
Таблица 13
Таксономическое положение сгруппированных гидробионтов
Botryococcus brauni и Diplosphaera mucosa
| Domain | Eukaryota | Eukaryota |
| Kingdom | Plantae | Plantae |
| Phylum | Chlorophyta | Chlorophyta |
| Class | Trebouxiophyceae | Chlorophyceae |
| Order | Trebouxiales | Chaetophorales |
| Family | Botryococcaceae | Chaetophoraceae |
| Genus, Species | Botryococcus brauni | Diplosphaera mucosa |
Как видно из табл. 13 кластер 12 содержит организмы, близкие по таксономическому положению на уровне Phylum, и вид Diplosphaera mucosa может рассматриваться как индикатор o–b–сапробности с вероятностью 77 %.
Таким образом, по результатам исследования гипотетического хода эволюции гидробионтов озера Верхний Кабан по маркерному гену 18S рРНК проанализировано обобщённое молекулярное филогенетическое дерево, выделено 12 устойчивых кластеров с высоким бутстреп-значением (68 %–100 %), приведены 13 видов, которые могут рассматриваться как индикаторы разных зон сапробности на основе их кластеризации с существующими видами-индикаторами, близкими по молекулярно-генетическому эволюционному развитию и таксономическому положению.
ЗАКЛЮЧЕНИЕ
В результате проведённого исследования по маркерному гену 18S рРНК гидробионтов озера Верхний Кабан список видов-индикаторов расширился на 13 видов:
водоросли: Diplosphaera mucosa (o–b–сапробность), Mucidosphaerium pulchellum (p–a–сапробность), Dinobryon pediforme (b–мезосапробность), Gomphoneis minuta
(b–мезосапробность), Nitzschia amphibia (o–b–сапробность), Rhabdonema adriaticum
(o–b–сапробность), Synura mammillosa (b–o–сапробность), Cryptomonas ozolini
(b–мезосапробность);
инфузории: Astylozoon enriquesi (b–мезосапробность), Blepharocorys curvigula
(p–сапробность), Enchelys gasterosteus (p–сапробность), Monocoronella carnea
(a–b–мезосапробность), Plagiopogon loricatus (b–a–мезосапробность).
Большинство выявленных видов-индикаторов группируются возле
b–мезосапробности, что указывает на экологическое состояние озера Верхний Кабан как на загрязненное по оценкам экологов.
REFERENCES
- 1. Frolova L. L., Sverdrup A. E., Malanin S. Yu., Drevenskaya O. Yu., Husainov A. V. and Kharchenko A. M. Metagenome of hydrobionts of Kaban lakes of Kazan city: a species diversity analysis by marker genes, 216 p. (Kazan University Press, 2019).
- Sladeček V. System of water quality from biological point of view, Arch. Hydrobiol. Beih., Ergebn. Limnol., 7, 1 (1973).
- Frolova L. L. and Sverdrup A. E. Diversity of Verhniy Kaban Lake by 18S rRNA of hydrobionts on next-generation sequecing method, Biosc.Biotech.Res.Comm., 12 (5), 329 (2019).
- Kharchenko A., Sverdrup A. and Frolova L. Studying Bacterial diversity of Verhniy Kaban Lake by 16S rRNA analysis, Helix Journal., 9 (5), 5545 (2019).
- Kharchenko A., Sverdrup A. and Frolova L. The monitoring of Verkhniy Kaban lake by rbcL gene of freshwater organisms using next-generation sequencing, International Journal of Green Pharmacy, 12 (03), 756 (2018).
- Frolova L. L. and Sverdrup A. E. A method of saprobity identification of hydrobionts for ecological assessment of water reservoirs, Patent of Russia №2698651, 25 (2019).
- Lafontaine D. L. J. and Tollervey D. Ribosomal RNA, https://www.lafontainelab.com/wp-content/uploads/Lafontaine2001els.pdf.
- Saitou N. and Nei M. The neighbor-joining method: A new method for reconstructing phylogenetic trees, Molecular Biology and Evolution, 4, 406 (1987).
- Tamura K., Nei M. and Kumar S. Prospects for inferring very large phylogenies by using the neighbor-joining method, Proceedings of the National Academy of Sciences (USA, 2004), p. 11030.
- Nei M. and Kumar S. Molecular Evolution and Phylogenetic (Oxford University Press, 2000).
- Kumar S., Stecher G., Li M., Knyaz Ch. and Namura K. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms, Molecular Biology and Evolution, 35 (6), 1547 (2018).
- Felsenstein J. Confidence limits on phylogenies: An approach using the bootstrap, Evolution, 39 (4), 783 (1985).



